Una vasta búsqueda de la diferencia natural ha llevado a los científicos en el Instituto McGovern para la Investigación del Brain del MIT y al amplio Instituto de MIT y Harvard para descubrir sistemas antiguos con potencial para expandir la caja de herramientas de tiraje del genoma.
Estos sistemas, que los investigadores llaman sistemas TIGR (ARN de maestro entre espaciados en tándem), usan ARN para guiarlos a sitios específicos en el ADN. Los sistemas TIGR pueden reprogramarse para orientar cualquier secuencia de interés de ADN, y tienen módulos funcionales distintos que pueden realizar sobre el ADN dirigido. Encima de su modularidad, TIGR es muy compacto en comparación con otros sistemas guiados por ARN, como CRISPR, lo cual es una gran superioridad para administrarlo en un contexto terapéutico.
Estos hallazgos son reportado en sarta el 27 de febrero en la revista Ciencia.
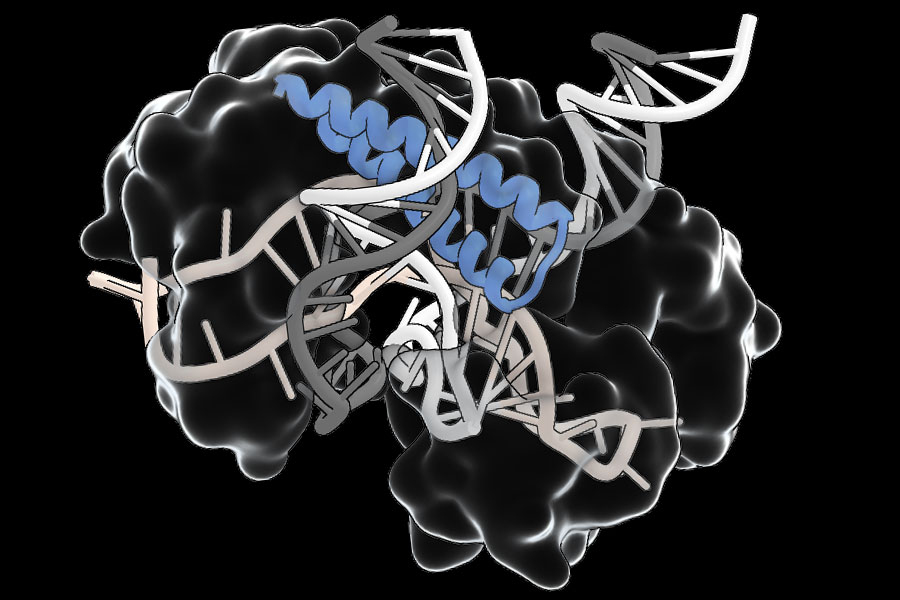
«Este es un sistema muy versátil guiado por ARN con muchas funcionalidades diversas», dice Feng Zhang, profesor de neurociencia de James y Patricia Poitras en el MIT, quien dirigió la investigación. Las proteínas asociadas a TIGR (TAS) que encontró el equipo de Zhang comparten un componente característico de unión a ARN que interactúa con una maestro de ARN que lo dirige a un sitio específico en el genoma. Algunos cortan el ADN en ese sitio, utilizando un segmento de corte de ADN adyacente de la proteína. Esa modularidad podría simplificar el explicación de herramientas, permitiendo a los investigadores permutar nuevas características aperos en proteínas Tas naturales.
«La naturaleza es sobrado increíble», dice Zhang, quien además es investigador del Instituto McGovern y del Instituto Médico Howard Hughes, miembro central del Broad Institute, profesor de cerebro y ciencias cognitivas e ingeniería biológica en el MIT, y codirector del K. Mújol Yang y Hock E. Centro Tanaprema de Tanapes Moleculares en el At Mit. «Tiene una gran cantidad de diferencia, y hemos estado explorando esa diferencia natural para encontrar nuevos mecanismos biológicos y aprovecharlos para diferentes aplicaciones para manipular procesos biológicos», dice. Anteriormente, el equipo de Zhang adaptó los sistemas CRISPR bacterianos en herramientas de tiraje de genes que han transformado la biología moderna. Su equipo además ha enemigo una variedad de proteínas programables, tanto de sistemas CRISPR como de más allá.
En su nuevo trabajo, para encontrar nuevos sistemas programables, el equipo comenzó a cero en una característica estructural de la proteína CRISPR-CAS9 que se une a la maestro de ARN de la enzima. Esa es una característica esencia que ha convertido a Cas9 en una útil tan poderosa: «Estar guiado por ARN hace que sea relativamente manejable reprogramarse, porque sabemos cómo se une el ARN a otro ADN u otro ARN», explica Zhang. Su equipo registró cientos de millones de proteínas biológicas con estructuras conocidas o predichas, buscando cualquiera que compartiera un dominio similar. Para encontrar proteínas relacionadas más distantemente, utilizaron un proceso iterativo: de Cas9, identificaron una proteína llamamiento IS110, que otros habían demostrado que otros se unían al ARN. Luego se centraron en las características estructurales de IS110 que permiten la unión de ARN y repitieron su búsqueda.
En este punto, la búsqueda había aumentado tantas proteínas distantemente relacionadas que el equipo recurrieron a la inteligencia industrial para dar sentido a la tira. «Cuando está haciendo minería iterativa y profunda, los éxitos resultantes pueden ser tan diversos que son difíciles de analizar utilizando métodos filogenéticos unificado, que se basan en la secuencia conservada», explica Guilhem Faure, un biólogo computacional en el laboratorio de Zhang. Con un maniquí de estilo conspicuo de proteínas, el equipo pudo agrupar las proteínas que habían enemigo en grupos de acuerdo con sus probables relaciones evolutivas. Un reunión apartó del resto, y sus miembros fueron particularmente intrigantes porque estaban codificados por genes con secuencias repetitivas espaciadas regularmente que recuerdan a un componente esencial de los sistemas CRISPR. Estos fueron los sistemas TIGR-TAS.
El equipo de Zhang descubrió más de 20,000 proteínas TA diferentes, principalmente ocurriendo en virus que infectan bacterias. Las secuencias internamente de la región repetitiva de cada gen, sus matrices TIGR, codifican una maestro de ARN que interactúa con la parte de unión a ARN de la proteína. En algunos, la región de unión a ARN está adyacente a una parte de corte de ADN de la proteína. Otros parecen unirse a otras proteínas, lo que sugiere que podrían ayudar a dirigir esas proteínas a objetivos de ADN.
Zhang y su equipo experimentaron con docenas de proteínas TAS, lo que demuestra que algunos pueden programarse para hacer cortes dirigidos al ADN en las células humanas. Mientras piensan en desarrollar sistemas TIGR-TAS en herramientas programables, las características alientan a los investigadores que podrían hacer que esas herramientas sean particularmente flexibles y precisas.
Señalan que los sistemas CRISPR solo pueden dirigirse a segmentos de ADN que están flanqueados por motivos cortos conocidos como PAM (motivos adyacentes de Protospacer). Las proteínas TigR Tas, en contraste, no tienen tal requisito. «Esto significa teóricamente, cualquier sitio en el genoma debería ser objetivo», dice el asesor estudiado Rhiannon Macrae. Los experimentos del equipo además muestran que los sistemas TIGR tienen lo que Faure claridad un «sistema de doble maestro», que interactúa con uno y otro hilos de la doble hélice de ADN a casa en sus secuencias objetivo, lo que debería certificar que actúen solo donde están dirigidos por su maestro de ARN. Encima, las proteínas TAS son compactas, una cuarta parte del tamaño CAS9, en promedio, haciéndolas más fáciles de entregar, lo que podría exceder un obstáculo importante para el despliegue terapéutico de herramientas de tiraje de genes.
Emocionado por su descubrimiento, el equipo de Zhang ahora está investigando el papel natural de los sistemas TIGR en los virus, así como cómo pueden adaptarse para la investigación o la terapéutica. Han determinado la estructura molecular de una de las proteínas TA que encontraron en células humanas, y utilizarán esa información para mandar sus esfuerzos para hacerlo más valioso. Encima, señalan las conexiones entre los sistemas TIGR-TAS y ciertas proteínas de procesamiento de ARN en células humanas. «Creo que hay más para estudiar en términos de cuáles pueden ser algunas de esas relaciones, y puede ayudarnos a comprender mejor cómo se usan estos sistemas en los humanos», dice Zhang.
Este trabajo fue apoyado por la Fundación Helen Hay Whitney, el Instituto Médico Howard Hughes, K. Mújol Yang y Hock E. Tan Center for Molecular Therapeutics, Broad Institute Programmable Therapeutics Gift Donors, Pershing Square Foundation, William Ackman, Neri Oxman, The Phillips Family, J. y P. Poitras y la Fundación Baritable BT.
