Las proteínas son los caballos de batalla que mantienen nuestras células en funcionamiento, y hay muchos miles de tipos de proteínas en nuestras células, cada uno realizando una función especializada. Los investigadores han sabido durante mucho tiempo que la estructura de una proteína determina lo que puede hacer. Más recientemente, los investigadores están llegando a apreciar que la ubicación de una proteína todavía es crítica para su función. Las células están llenas de compartimentos que ayudan a organizar sus muchos habitantes. Anejo con los orgánulos conocidos que adornan las páginas de los libros de texto de biología, estos espacios todavía incluyen una variedad de compartimentos dinámicos sin membrana que concentran ciertas moléculas para realizar funciones compartidas. Por lo tanto, aprender dónde se localiza una proteína determinada y con quién se localiza conjuntamente para comprender mejor que la proteína y su papel en la célula sana o enferma, pero los investigadores han carecido de una forma sistemática de predecir esta información.
Mientras tanto, la estructura de proteínas se ha estudiado durante más de medio siglo, que culminó en la utensilio de inteligencia químico Alfafold, que puede predecir la estructura de proteínas a partir del código de aminoácidos de una proteína, la sujeción directo de bloques de construcción internamente de ella que se pliega para crear su estructura. Alfafold y modelos como este se han convertido en herramientas ampliamente utilizadas en la investigación.
Las proteínas todavía contienen regiones de aminoácidos que no se pliegan en una estructura fija, sino que son importantes para ayudar a las proteínas a unir compartimentos dinámicos en la célula. El profesor del MIT Richard Young y sus colegas se preguntaron si el código en esas regiones podría estar de moda para predecir la ubicación de proteínas de la misma modo que otras regiones se usan para predecir la estructura. Otros investigadores han descubierto algunas secuencias de proteínas que codifican la ubicación de proteínas, y algunos han comenzado a desarrollar modelos predictivos para la ubicación de proteínas. Sin secuestro, los investigadores no sabían si la ubicación de una proteína en cualquier compartimento dinámico podría predecirse en función de su secuencia, ni tenían una utensilio comparable para alfafold para predecir la ubicación.
Ahora, Young, todavía miembro del Instituto Whitehead para la Investigación Biológica; Young Lab Postdoc Henry Kilgore; Regina Barzilay, profesora distinguida de la AI y la vitalidad del MIT en el Unidad de Ingeniería Eléctrica e Informática e Investigador Principal en el Laboratorio de Informática e Inteligencia Químico (CSAIL); Y los colegas han construido un maniquí de este tipo, que llaman protgps. En un artículo publicado en 6 de febrero en el diario Cienciacon los primeros autores Kilgore y los estudiantes graduados del laboratorio de Barzilay, Itamar Chinn, Peter Mikhael e Ilan Mitnikov, el equipo interdisciplinario debuta su maniquí. Los investigadores muestran que PROGPP puede predecir cuáles de los 12 tipos conocidos de compartimentos se localizarán una proteína, así como si una mutación asociada a la enfermedad cambiará esa ubicación. Encima, el equipo de investigación desarrolló un operación generativo que puede diseñar nuevas proteínas para localizarse en compartimentos específicos.
«Espero que este sea un primer paso alrededor de una plataforma poderosa que permita a las personas que estudian proteínas hacer su investigación», dice Young, «y que nos ayuda a comprender cómo los humanos se desarrollan en los organismos complejos que son, cómo las mutaciones interrumpen aquellos que Procesos naturales y cómo ocasionar hipótesis terapéuticas y diseñar medicamentos para tratar la disfunción en una célula ”.
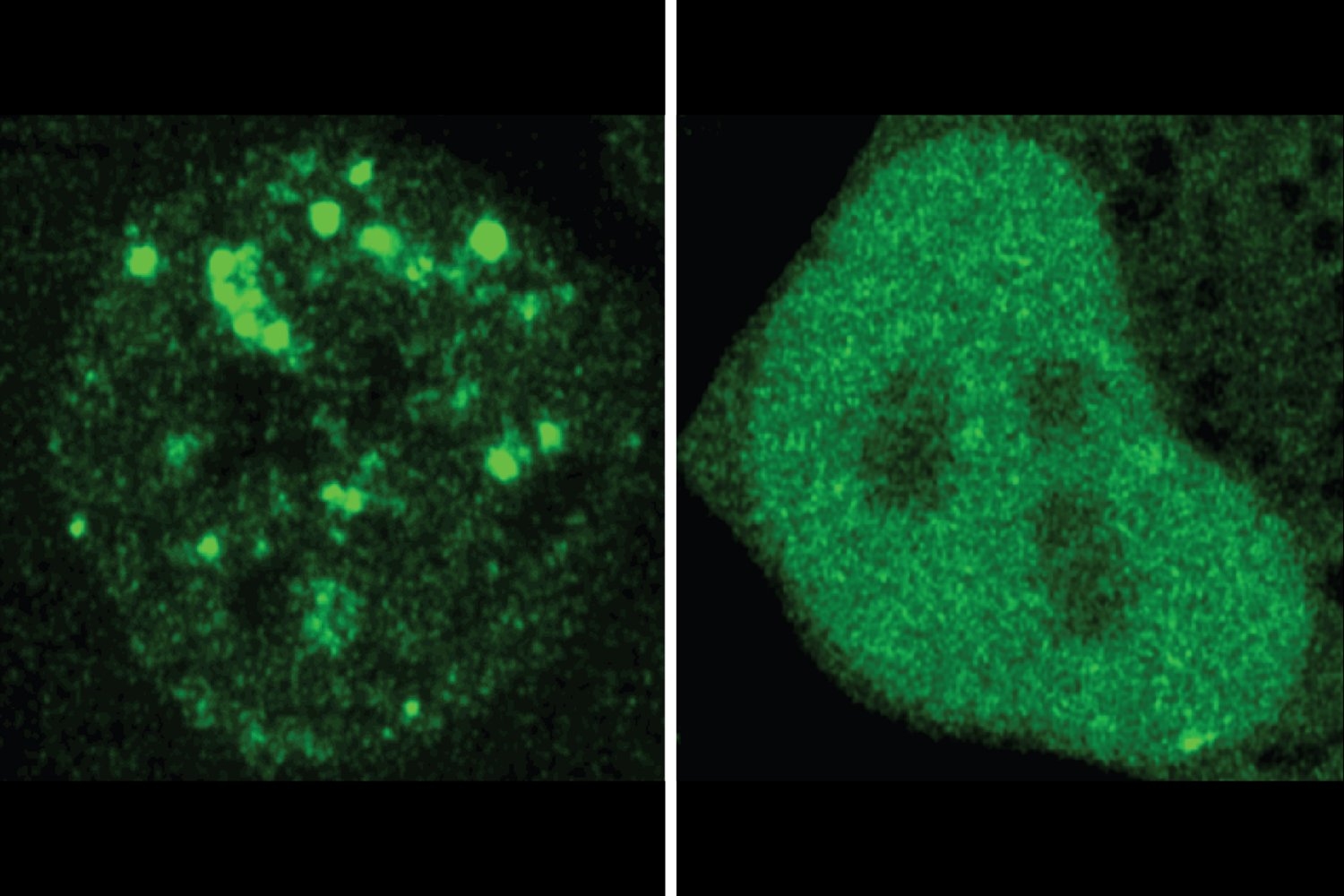
Los investigadores todavía validaron muchas de las predicciones del maniquí con pruebas experimentales en células.
«Verdaderamente me emocionó poder tener lugar del diseño computacional hasta probar estas cosas en el laboratorio», dice Barzilay. “Hay muchos documentos emocionantes en esta ámbito de IA, pero el 99.9 por ciento de ellos nunca se prueban en sistemas reales. Gracias a nuestra colaboración con el laboratorio pipiolo, pudimos probar y en realidad cultivarse qué tan perfectamente está haciendo nuestro operación «.
Desarrollar el maniquí
Los investigadores capacitaron y probaron PROGPS en dos lotes de proteínas con localizaciones conocidas. Descubrieron que podía predecir correctamente dónde las proteínas terminan con ingreso precisión. Los investigadores todavía probaron qué tan perfectamente PROGGP podría predecir cambios en la ubicación de proteínas basadas en mutaciones asociadas a la enfermedad internamente de una proteína. Se ha enemigo que muchas mutaciones, cambios en la secuencia de un gen y su proteína correspondiente, contribuyen o causan enfermedad en función de los estudios de asociación, pero las formas en que las mutaciones conducen a los síntomas de la enfermedad siguen siendo desconocidas.
Descubrir el mecanismo de cómo una mutación contribuye a la enfermedad es importante porque los investigadores pueden desarrollar terapias para fijar ese mecanismo, avisar o tratar la enfermedad. Los jóvenes y sus colegas sospecharon que muchas mutaciones asociadas a la enfermedad podrían contribuir a la enfermedad al cambiar la ubicación de proteínas. Por ejemplo, una mutación podría hacer que una proteína no pueda unirse a un compartimento que contenga socios esenciales.
Probaron esta hipótesis alimentando a Protgos más de 200,000 proteínas con mutaciones asociadas a la enfermedad, y luego pidieron que predice dónde esas proteínas mutadas localizarían y medirían cuánto cambió su predicción para una proteína dada de la interpretación regular a la mutada. Un gran cambio en la predicción indica un cambio probable en la ubicación.
Los investigadores encontraron muchos casos en los que una mutación asociada a la enfermedad parecía cambiar la ubicación de una proteína. Probaron 20 ejemplos en las células, utilizando fluorescencia para comparar en qué parte de la célula una proteína regular y la interpretación mutada terminó. Los experimentos confirmaron las predicciones de ProTGPS. En total, los hallazgos respaldan la sospecha de los investigadores de que la ubicación errónea puede ser un mecanismo de enfermedad subestimado y demuestra el valía de PROGGP como una utensilio para comprender la enfermedad e identificar nuevas vías terapéuticas.
«La celda es un sistema tan complicado, con tantos componentes y redes complejas de interacciones», dice Mitnikov. «Es muy interesante pensar que con este enfoque, podemos perturbar el sistema, ver el resultado de eso y, por lo tanto, impulsar el descubrimiento de mecanismos en la célula o incluso desarrollar terapéuticas basadas en eso».
Los investigadores esperan que otros comiencen a usar PROGPS de la misma modo que usan modelos estructurales predictivos como Alfafold, avanzando varios proyectos sobre la función de proteínas, la disfunción y la enfermedad.
Ir más allá de la predicción a una procreación novedosa
Los investigadores estaban entusiasmados con los posibles usos de su maniquí de predicción, pero todavía querían que su maniquí fuera más allá de predecir las localizaciones de las proteínas existentes y les permitiera diseñar proteínas completamente nuevas. El objetivo era que el maniquí compensara secuencias de aminoácidos completamente nuevas que, cuando se forman en una célula, se localizarían en una ubicación deseada. Gestar una proteína nueva que en sinceridad puede ganar una función, en este caso, la función de localizarse en un compartimento celular específico, es increíblemente difícil. Para mejorar las posibilidades de éxito de su maniquí, los investigadores restringieron su operación para diseñar solo proteínas como las que se encuentran en la naturaleza. Este es un enfoque comúnmente utilizado en el diseño de fármacos, por razones lógicas; La naturaleza ha tenido miles de millones de abriles para determinar qué secuencias de proteínas funcionan perfectamente y cuáles no.
Adecuado a la colaboración con el laboratorio pipiolo, el equipo de formación maquinal pudo probar si su creador de proteínas funcionaba. El maniquí tuvo buenos resultados. En una ronda, generó 10 proteínas destinadas a localizarse en el nucleolo. Cuando los investigadores probaron estas proteínas en la célula, descubrieron que cuatro de ellas fuertemente localizadas en el nucleolo, y otros todavía pueden suceder tenido ligeros sesgos alrededor de esa ubicación.
«La colaboración entre nuestros laboratorios ha sido tan generativa para todos nosotros», dice Mikhael. “Hemos aprendido cómo conversar los idiomas de los demás, en nuestro caso aprendimos mucho sobre cómo funcionan las células y al tener la oportunidad de probar experimentalmente nuestro maniquí, hemos podido descubrir qué debemos hacer para hacer en realidad El maniquí funciona y luego lo hace funcionar mejor «.
Ser capaz de ocasionar proteínas funcionales de esta modo podría mejorar la capacidad de los investigadores para desarrollar terapias. Por ejemplo, si un medicamento debe interactuar con un objetivo que se localiza internamente de un cierto compartimento, los investigadores podrían usar este maniquí para diseñar un medicamento para colocar todavía allí. Esto debería hacer que el medicamento sea más efectivo y disminuir los género secundarios, ya que el medicamento pasará más tiempo comprometiendo con su objetivo y menos tiempo interactuando con otras moléculas, causando género fuera del objetivo.
Los miembros del equipo de formación maquinal están entusiasmados con la posibilidad de usar lo que han aprendido de esta colaboración para diseñar proteínas novedosas con otras funciones más allá de la ubicación, lo que ampliaría las posibilidades de diseño terapéutico y otras aplicaciones.
«Muchos documentos muestran que pueden diseñar una proteína que se pueda expresar en una célula, pero no que la proteína tenga una función particular», dice Chinn. “En sinceridad teníamos un diseño de proteínas funcionales y una tasa de éxito relativamente noble en comparación con otros modelos generativos. Eso es en realidad emocionante para nosotros, y poco en lo que nos gustaría construir «.
Todos los investigadores involucrados ven a Protgps como un manifestación emocionante. Anticipan que su utensilio se utilizará para cultivarse más sobre los roles de la ubicación en la función de proteínas y la ubicación errónea en la enfermedad. Encima, están interesados en expandir las predicciones de ubicación del maniquí para incluir más tipos de compartimentos, probar más hipótesis terapéuticas y diseñar proteínas cada vez más funcionales para terapias u otras aplicaciones.
«Ahora que sabemos que este código de proteína para la ubicación existe, y que los modelos de formación maquinal pueden dar sentido a ese código e incluso crear proteínas funcionales utilizando su deducción, que abre la puerta a tantos estudios y aplicaciones potenciales», dice Kilgore.
